Un nouvel outil d’édition génétique cible les mutations avec une précision inégalée

Résumé: Un outil révolutionnaire appelé Helicase-Assisted Continu Editing (HACE) permet aux scientifiques de créer des mutations génétiques précises dans des gènes spécifiques sans affecter le reste du génome. En combinant les enzymes hélicase avec la technologie CRISPR, HACE introduit des mutations dans des séquences d’ADN ciblées, améliorant ainsi notre capacité à étudier les fonctions des gènes et les mécanismes des maladies. L’outil a déjà identifié des mutations de résistance aux médicaments dans des gènes liés au cancer et des défauts d’épissage dans les cancers du sang, démontrant ainsi son potentiel de découverte thérapeutique.
Faits clés:
- HACE introduit des mutations dans des gènes spécifiques, laissant le reste du génome intact.
- Il a identifié des mutations liées à la résistance aux médicaments anticancéreux et aux défauts d’épissage.
- Cet outil pourrait révolutionner la découverte thérapeutique et la recherche génomique.
Source: Harvard
Les mutations génétiques ont des conséquences à la fois bonnes et mauvaises – de la résistance à des maladies comme le diabète à la susceptibilité à certains cancers.
Afin d’étudier ces mutations, les scientifiques doivent les introduire directement dans les cellules humaines. Mais modifier les instructions génétiques à l’intérieur des cellules est complexe. Le génome humain comprend 3 milliards de paires de bases d’ADN réparties sur des dizaines de milliers de gènes.
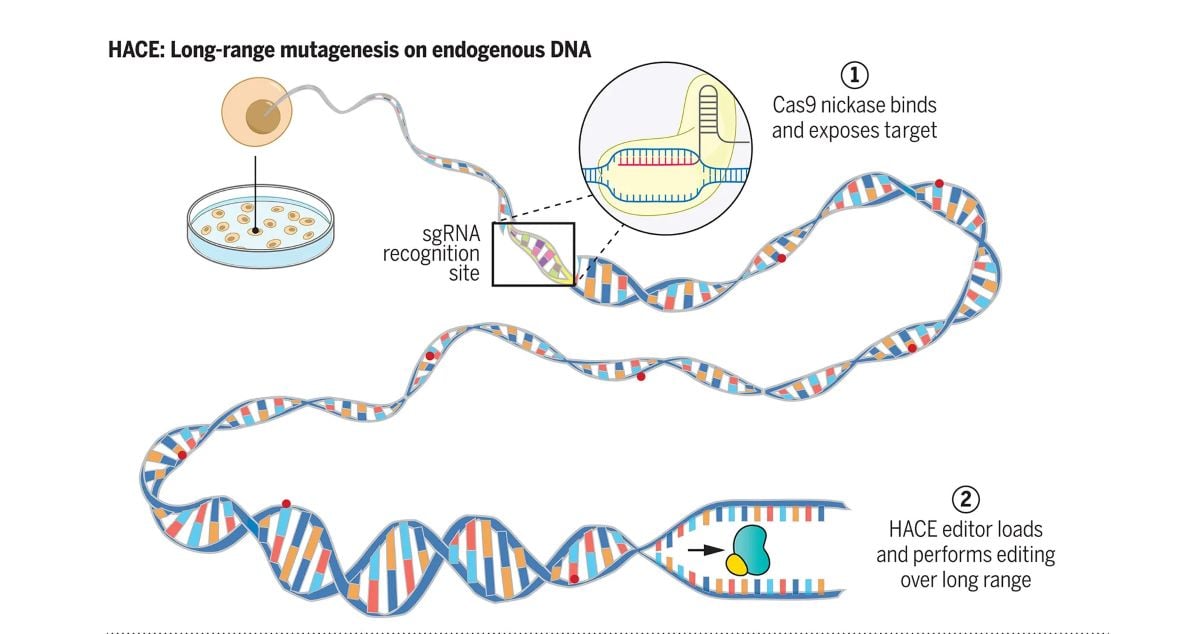
À cette fin, les chercheurs de Harvard ont créé un outil qui leur permet de créer rapidement des mutations uniquement dans des gènes d’intérêt particuliers sans perturber le reste du génome. Décrit dans Scienceleur outil, appelé Helicase-Assisted Continu Editing (HACE), peut être déployé sur des régions prédéterminées du génome dans des cellules vivantes intactes.
« Le développement d’outils comme celui-ci marque un pas en avant significatif dans notre capacité à exploiter l’évolution directement au sein des cellules humaines », a déclaré le premier auteur Xi Dawn Chen, étudiante à la Griffin Graduate School of Arts and Sciences étudiant la biologie synthétique au Département de cellules souches et Biologie régénérative.
« En permettant une mutagenèse ciblée dans des parties spécifiques du génome, cet outil ouvre la porte à la création d’enzymes et de traitements auparavant hors de portée. »
Contrairement aux méthodes actuelles de mutagenèse, qui impliquent l’insertion de copies supplémentaires de gènes ou la mutation simultanée de nombreux gènes différents, HACE offre l’avantage d’être dirigé vers des emplacements – comme se rendre à une adresse spécifique plutôt qu’à un quartier. La nouvelle bio-ingénierie de l’équipe consiste à combiner une hélicase, qui est une enzyme qui « décompresse » naturellement l’ADN, avec une enzyme d’édition de gènes.
Ils utilisent ensuite la technologie d’édition de gènes CRISPR-Cas9 pour guider la paire de protéines vers le gène qu’ils souhaitent muter. Lorsque l’hélicase décompresse l’ADN, elle introduit des mutations uniquement dans cette séquence génétique.
« HACE combine la précision de CRISPR avec la capacité de modifier de longues portions d’ADN, ce qui en fait un outil puissant pour une évolution ciblée », a expliqué l’auteur principal Fei Chen, professeur adjoint au Département de cellules souches et de biologie régénérative et membre du Broad Institute.
Pour démontrer la puissance de l’outil en laboratoire, les scientifiques l’ont utilisé pour identifier des mutations de résistance aux médicaments dans un gène appelé MEK1, que les traitements contre le cancer ciblent souvent mais échouent fréquemment parce que les cellules malades mutent les mécanismes de résistance.
En utilisant HACE, l’équipe a séquencé uniquement ces gènes mutés et identifié plusieurs changements uniques associés à la résistance aux médicaments anticancéreux comme le trametinib et le sélumétinib, offrant ainsi un aperçu de la manière dont les mutations affectent les performances des médicaments.
Ils ont également examiné comment les mutations de SF3B1, un gène impliqué dans un processus biomoléculaire appelé épissage de l’ARN, affectent l’assemblage de l’ARN. Les mutations de ce gène sont courantes dans les cancers du sang, mais on ne sait pas exactement quelles mutations sont à l’origine des défauts d’épissage ; avec HACE, l’équipe a pu facilement identifier ces changements.
Et en partenariat avec le laboratoire de Bradley Bernstein de la Harvard Medical School et du Dana-Farber Cancer Institute, les chercheurs ont également utilisé l’outil pour mieux comprendre comment les changements dans une région régulatrice de l’ADN affectent la production d’une protéine dans les cellules immunitaires reconnues comme une cible potentielle du cancer. immunothérapies.
Bernstein a déclaré que des outils comme HACE pourraient un jour permettre des modifications massives de séquences régulatrices de gènes qui pourraient ensuite être couplées à un calcul d’apprentissage profond pour le déchiffrement. « On peut imaginer de nombreuses nouvelles opportunités thérapeutiques qui impliquent des modifications ou un réglage précis de ces séquences régulatrices pour » corriger « l’activité des gènes et améliorer la maladie », a déclaré Bernstein.
Financement: Cette recherche a été soutenue par plusieurs sources, notamment les National Institutes of Health, le Broad Institute et le Harvard Stem Cell Institute.
A propos de cette actualité de la recherche en génétique
Auteur: Anne J. Manning
Source: Harvard
Contact: Anne J. Manning – Harvard
Image: L’image est créditée à Neuroscience News
Recherche originale : Accès fermé.
« Édition continue assistée par hélicase pour la mutagenèse programmable des génomes endogènes» par Xi Dawn Chen et al. Science
Abstrait
Édition continue assistée par hélicase pour la mutagenèse programmable des génomes endogènes
INTRODUCTION
L’un des défis fondamentaux de la génomique consiste à déterminer l’impact des trois milliards de bases du génome humain sur la fonction des protéines et la régulation des gènes. Ainsi, un objectif essentiel est de développer des stratégies permettant de mutagéniser les séquences génomiques de manière systématique et à haut débit.
En particulier, une mutagenèse ciblée de loci génomiques uniques pourrait imiter le processus d’évolution naturel pour révéler des relations séquence-structure, des phénotypes de gain et de perte de fonction et des mutations coopératives. Cependant, il n’existe aucune méthode permettant d’effectuer une mutagenèse continue dans des régions ciblées du génome endogène des cellules de mammifères.
RAISONNEMENT
Nous avons cherché à développer un outil permettant de réaliser une mutagenèse ciblée sur le génome endogène des mammifères. En ce qui concerne la nature, nous avons observé que les hélicases sont des enzymes hautement processives capables de traverser de vastes régions génomiques. Certaines hélicases, notamment celles impliquées dans la réparation des dommages à l’ADN, peuvent charger et commencer à dérouler l’ADN au niveau des régions d’ADN simple brin du génome.
Nous avons estimé que de telles hélicases pourraient être utilisées pour une mutagenèse ciblée à longue portée lorsqu’elles sont fusionnées à une enzyme désaminase. La construction de fusion et son intervalle d’hypermutation pourraient ensuite être ciblés de manière programmable, via des ARN à guide unique (sgRNA), vers des régions génomiques spécifiques à l’aide d’une nickase Cas9. L’événement de déroulement directionnel et à longue portée de l’ADN par l’hélicase recrutée générera alors des mutations aléatoires dans la région.
RÉSULTATS
Nous avons conçu une plate-forme appelée édition continue assistée par hélicase (HACE), qui combine l’édition à longue portée de loci entiers avec la programmabilité de séquence inhérente aux outils d’édition génétique CRISPR. HACE utilise CRISPR-Cas9 pour diriger le chargement d’une fusion hélicase-désaminase pour une hypermutation ciblée de la séquence génomique en aval. HACE a obtenu une désamination spécifique au locus sur plus de 1 000 nucléotides, les mutations s’accumulant continuellement au fil du temps.
Nous avons en outre évalué les prototypes HACE incorporant diverses hélicases, variantes Cas9 et désaminases, montrant qu’ils ont des taux et des plages d’édition réglables. Nous avons également montré que HACE peut être multiplexé pour cibler plusieurs régions génomiques avec un nombre minimal d’ARN guides. Nous avons ensuite appliqué HACE dans des contextes génomiques codants et non codants pour disséquer fonctionnellement les mutations endogènes conférant une résistance aux médicaments, des modifications de l’activité enzymatique et une modification de la fonction des éléments cis-régulateurs.
Dans l’espace de codage, nous avons identifié des variantes qui conduisent à une résistance aux médicaments inhibiteurs de la protéine kinase kinase 1 (MEK1) activée par un mitogène, ainsi que des variantes de SF3B1, un facteur d’épissage, qui conduisent à une utilisation alternative du site d’épissage en 3 ‘. En ce qui concerne les régions régulatrices, nous avons défini des variantes artificielles fonctionnelles dans les régions amplificatrices de CD69 et identifié des bases et des motifs spécifiques qui médient l’impact des facteurs de transcription RUNX sur la régulation de CD69.
HACE résout deux limitations rencontrées par les écrans d’édition de bases conventionnels : l’exigence d’un motif adjacent au protospacer NGG dans la séquence de reconnaissance du sgARN et l’apparition de mutations spectateurs qui peuvent créer des liaisons artificielles et confondre les résultats du dépistage. La longue plage d’édition de HACE peut également découvrir des effets combinatoires et des interactions entre plusieurs mutations distantes sur un locus.
CONCLUSION
HACE rend possible la diversification continue, programmable et à longue portée des génomes endogènes de mammifères. Nous envisageons que HACE élargira considérablement la boîte à outils de la génomique fonctionnelle et permettra la création de cartes séquence-fonction systématiques des génomes codants et non codants.
De plus, HACE peut être développé en un système d’évolution dirigée dans le génome endogène, permettant la sélection de séquences pour les fonctions souhaitées en biologie des mammifères.



